
Technology
Background
がんや希少疾患に新たな治療選択肢を
がんや希少疾患の原因の多くは遺伝子変異に関係しています。これまでの低分子医薬品や抗体医薬品では、分子の立体構造に依存するため、標的となる全てのタンパク質に対応することは難しいとされています。現在においても、遺伝子変異に関係する疾患の多くは、治療薬が存在しない状況です。より効果的な医療を提供するためには、セントラルドグマ(「DNA→mRNA→タンパク質」の順に伝達されるという、分子生物学の概念)の中心に入り込む必要があります。それが核酸医薬です。
核酸医薬品は目的とする1つの標的遺伝子のみを特異的に抑制することができるので、「第三の医薬品」として近年、世界中で注目を集めています。
現在、製品化されている核酸医薬は主にsiRNAとアンチセンスで、そのうちsiRNA医薬品は5つ存在します。最近では年1-2製品が市場投入されている状況で、今後も製品拡大が見込まれています。siRNAの作用機序はRNA干渉(RNAi:後述)という細胞に元来備わっている機能を利用しており、これにより効果的にmRNAの抑制を行っています。
RNAi
ノーベル賞受賞技術RNAi(RNA interference)とは
RNAiとは2006年にノーベル生理学・医学賞を受賞した、二本鎖RNAと相補的な配列を持つmRNAが特異的に分解される現象で、siRNAもこの作用機序を利用しています。siRNAと相補的な配列を持つmRNAが分解されることによって、最終的に標的タンパク質の発現量が減少する作用をもたらします。
詳細は、以下の流れとなります。
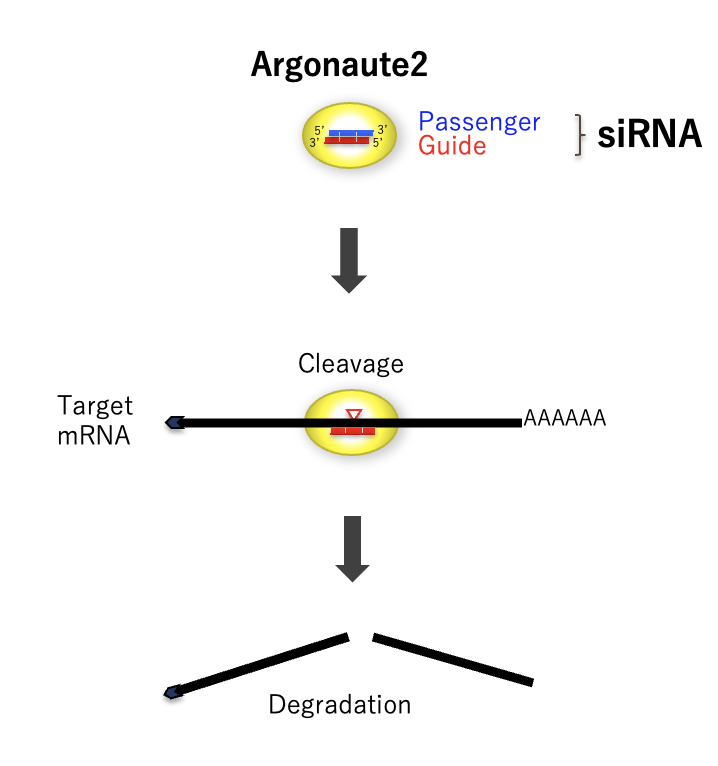
① siRNAが細胞質に取り込まれます。
② siRNAはArgonaute2(AGO2)タンパク質にローディングされます。
③ siRNAはAGO2上で、二本鎖のうち標的mRNAと対合する側のRNA鎖(ガイド鎖)の5′末端の塩基がAGO2のポケット構造に固定され、ガイド鎖と相補的な配列を有するもう一方のRNA鎖(パッセンジャー鎖)が除去されます。
④ AGO2上で、siRNAガイド鎖と相補的な配列をもつ標的mRNAが対合し、AGO2によって切断されます。切断された標的mRNAは分解され、タンパク質の発現量が抑制されます。
Problem
核酸医薬品の現状と課題
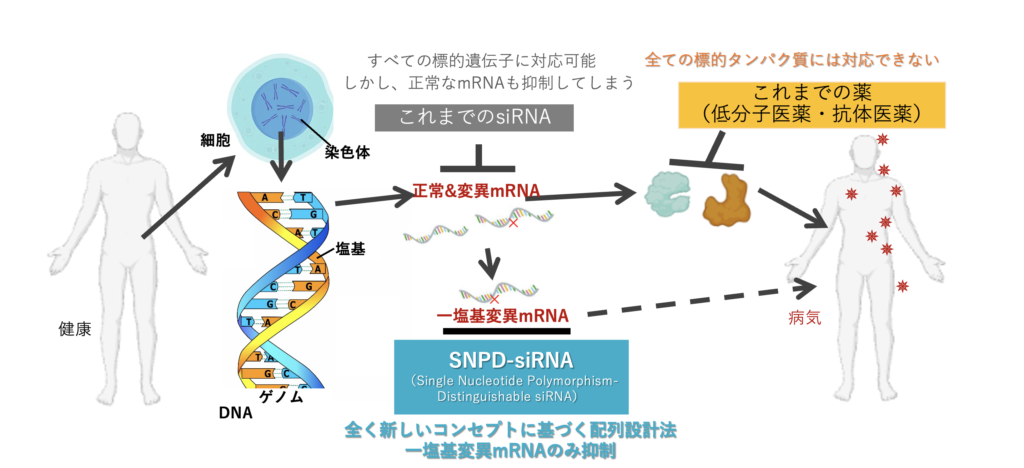
核酸医薬品、とくにsiRNAは化学合成が可能であること、配列設計の法則が存在していることが魅力的とされています。さらに、副作用がほとんど出ず、数ヶ月に一度の注射で治療を行うことが可能となってきています。この分野のリーディングカンパニーである米国Alnylam Pharmaceuticals社(以下、Alnylam社)は、既に承認されている全てのsiRNA医薬品の開発を行っています。いずれも肝細胞をターゲットとした疾患となります。従来のsiRNA配列設計技術は、標的遺伝子のみを特異的に抑制することができますが、mRNAにおける一塩基変異の有無までは制御できません。そのため、Alnylam社は正常、変異mRNAの両方を抑制しても影響が出ない標的遺伝子・疾患ターゲットを選んで創薬を行う、特徴的なアプローチを取っています。
Our Solution
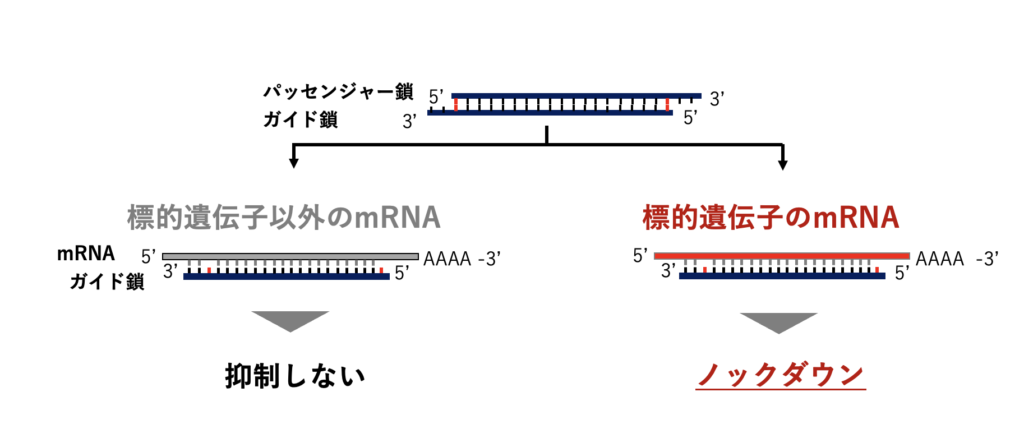
ANRisは東京大学 程久美子准教授が発明した、一塩基変異mRNAのみを選択的に抑制可能なsiRNA創薬における配列設計をコア技術としています。
現状の配列設計法では一塩基変異を含まない正常なmRNAも抑制してしまうことが課題でした。がんや多くの遺伝性疾患においては、標的遺伝子のうち正常なmRNAを抑制してしまうと副作用が生じ、治療の際に患者さんに負担を強いることとなってしまいます。そこで産み出されたのが、SNPD-siRNA(Single nucleotide polymorphism-distinguishable siRNA)です。本発明は一塩基変異を含むmRNAのみを特異的に抑制することができる革新的な配列設計法で、長年の様々な実験的な試行錯誤から体系化がなされました。
Proof of Concept
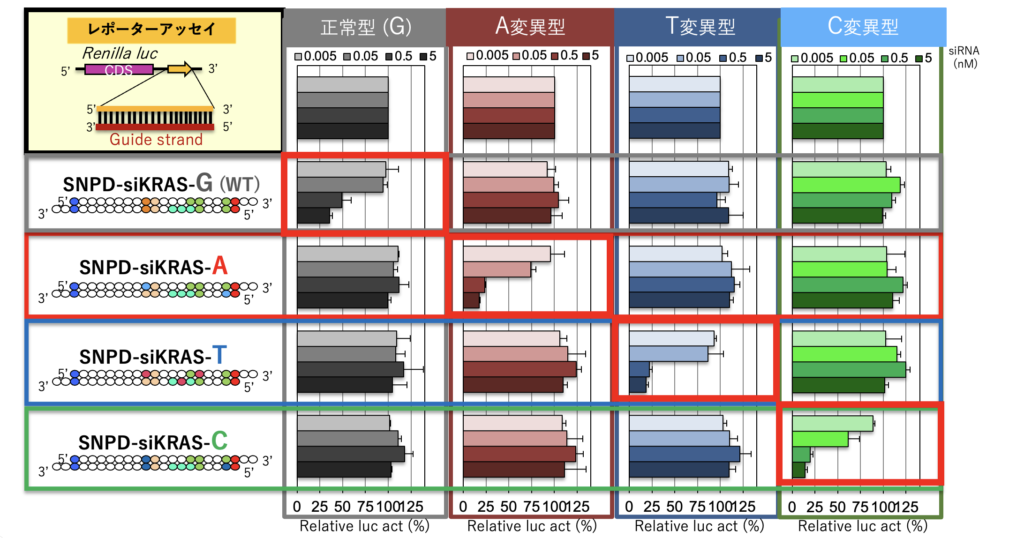
SNPD-siRNA技術の実証を行うため、がん発生・進行に直接的な役割を果たす「ドライバー遺伝子」の1つであるKRASに着目しました。KRAS(35塩基目)に対応するSNPD-siRNAを設計・合成し遺伝子発現制御解析(レポーターアッセイ)を行ったところ、一塩基の違いを識別して特異的に抑制するRNA干渉効果が認められました。また、膵臓がん由来細胞の平面培養、及びゼノグラフトモデル(ヒト由来のがん細胞を免疫不全マウスへ移植するモデル)マウスを用いた場合においても有効性を確認しております。
Reference
程久美子准教授らによる、siRNAに関わる主要論文
・Kobayashi Y, et al. ACS Omega, 7, 2398-2410, 2021.
・Iribe H, et al. ACS Omega 2, 2055-2064, 2017.
・Naito Y, et al. BMC Bioinformatics 10, 392, 2009.
・Ui-Tei K, et al. Nucleic Acids Res. 36, 7100-7109, 2008.
・Ui-Tei K, et al. Nucleic Acids Res. 36, 2136-2151, 2008.
・Naito Y, et al. Nucleic Acids Res. 32, W124-129 , 2004.
・Ui-Tei K, et al. Nucleic Acids Res. 32, 936-948, 2004.
・Doi N. et al. Curr. Biol. 13, 41-46, 2003.
